
GE@Lyon
Parcours Génomique environnementale
Le M2 parcours GE « Génomique environnementale » a accueilli sa première promotion d’étudiants dans sa nouvelle version à la rentrée 2022. Vous trouverez ci-dessous toutes les informations nécessaires pour découvrir la nouvelle offre de formation, son fonctionnement, les modalités d’inscription et les débouchés.

Responsables : Annabelle Haudry, Tristan Lefébure & Mylène Hugoni
Laboratoire d’écologie des hydrosystèmes naturels et anthropisés (LEHNA)
UMR 5023
Laboratoire de Biométrie et Biologie Évolutive (LBBE)
UMR CNRS 5558
contact: annabelle.haudry@univ-lyon1.fr / tristan.lefebure@univ-lyon1.fr / mylene.hugoni@univ-lyon1.fr
Formation
Objectifs de la formation
Nous savons maintenant que notre compréhension de la biodiversité reste très parcellaire à l’échelle globale. En effet, des environnements sont difficilement accessibles (eg. fonds océaniques), des branches entières du vivant restent inexplorées (eg. archées), la plupart des associations symbiotiques sont peu connues (eg. microbiomes), et même pour les pans de biodiversité les mieux connus, nos outils de suivi de la biodiversité sont mal adaptés face aux défis des changements actuels et à venir. Dans ce cadre, l’essor récent de la génomique, et en particulier les possibilités de séquencage haut-débit, offre une large palette d’outils et de méthodes pour décrire la biodiversité, étudier son fonctionnement et comprendre son évolution. L’objectif du parcours GE est de former des étudiant.e.s aux outils de la génomique pour répondre aux défis écologiques actuels.
Contenu pédagogique et organisation des cours
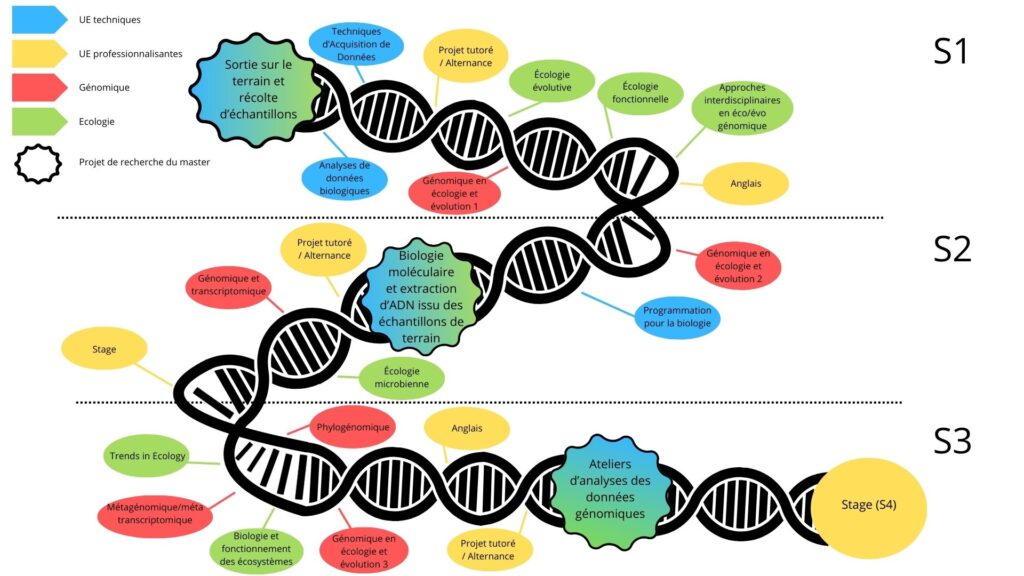
Master 1 - Semestre 1
Pour plus d’informations sur le premier semestre du M1, rendez-vous ici.
Master 1 - Semestre 2
Le semestre 2 renforce les compétences en génomique, en bioinformatique, en écologie microbienne, en biologie moléculaire (préparation et séquençage des échantillons collectés au S1); et enfin met les étudiant.es en situation d’autonomie sur un projet tutoré et un stage.
Approfondissement des connaissances en Génomique pour l’Ecologie et l’Evolution : dynamique d’évolution des génomes (impact de transferts horizontaux, effets de la recombinaison, insertion d’éléments mobiles) et reconstruction de l’histoire évolutive de populations.
Une UE de Biologie Moléculaire afin de développer des approches allant de la génomique à la métagénomique: préparation et séquençage des échantillons prélevés au S1.
Un enseignement spécialisé sur la Microbiologie Environnementale.
Une UE de Programmation pour la Biologie (apprentissage de python, accès à un serveur distant, manipulation de gros jeux de données), complétée par une UE de bioinformatique pour apprendre à traiter des données de Génomique et Transcriptomique.
Mise en autonomie sur un projet tutoré ainsi qu’un stage de 7 semaines.
Dynamique des génomes. Taille des génomes, éléments transposables, duplications.
Contribution des transfert horizontaux à l’évolution des génomes eucaryotes
Recombinaison, sélection et évolution du génome : conséquences de la recombinaison et de l’absence de recombinaison dans l’évolution du génome. Interaction entre recombinaison et sélection.
Reconstruction de l’histoire évolutive des populations, introduction au coalescent, impact de la sélection, tests de détection de sélection.
Responsable : Annabelle Haudry
L’objectif de cette unité d’enseignement est d’assurer aux biologistes une certaine autonomie en bioinformatique pour le développement d’outils spécifiques à leur problématique biologique et au traitement bioinformatique de leurs données.
Dans cette UE, les étudiant.e.s manipulent différents types de matrices et d’échantillons (ex. ADN environnementaux, échantillons d’eau ou de sol) pour développer des approches allant de la génomique à la métagénomique (ex. reconstruction de génomes mitochondriaux ou bactériens, description des communautés piscicoles par eDNA, caractérisation de communautés microbiennes environnementales). Les étudiant.e.s sélectionnent les approches de biologie moléculaire adaptées à leur question biologique / application, et réalisent les extractions, contrôles qualité et préparation de librairies de séquençage (Illumina et Nanopore). Dans le cas des librairies Nanopore, les étudiant.e.s réalisent eux-même le séquençage.
Ces approches seront développées avec une grande part d’autonomie, afin de préparer les étudiant.e.s au développement de protocoles de biologie moléculaire au cours de leurs stages et postes futurs.
Les données de séquençage acquises lors de cette UE seront analysées au S3 dans l’UE Ateliers d’Analyses en Génomique Environnementale.
Cette UE nécessite des prérequis en biologie moléculaire, qui sont enseignés dans l’UE TAD (S1).
Responsable : Clémentine François
Les différentes technologies d’acquisition de données génomiques et transcriptomiques, ainsi que leurs biais, seront présentées en lien avec des problématiques variées. L’accent sera mis sur les nouvelles technologies de séquençage (e.g. Illumina, Nanopore) et leur utilisation en génomique environnementale.
Les aspects technologiques seront mis en lien avec les spécificités biologiques dépendant de la nature des organismes étudiés (procaryotes ou eucaryotes). Seront abordées deux situations classiques d’assemblage de génome : i) un contexte de séquencage de novo (à partir de short et/ou de long reads) et ii) un contexte de reséquencage avec comparaison à un génome de référence (allant du mapping à l’analyse des polymorphismes découverts par SNP calling). Les différents types de contaminations dans les données de séquençage, leurs conséquences et leur détection seront abordées (TP). Les méthodes d’annotation structurale et fonctionnelle des génomes seront présentées et manipulées (e.g. détection de gènes, d’éléments régulateurs).
Les étudiants apprendront également à analyser des données transcriptomiques (assemblage, quantification d’exons, de jonctions, de gènes, estimation des taux d’expression). Le problème de la reconstruction de tous les transcrits alternatifs d’un gène sera étudié. L’analyse comparative des niveaux d’expression entre deux conditions expérimentales (traité / non traité, tissu1 /tissu2) sera l’occasion d’introduire les méthodes statistiques pour décider si un gène est différentiellement exprimé ou épissé.
Responsable : Annabelle Haudry
L’objectif de l’UE est l’acquisition des concepts théoriques et des méthodologies à l’interface de la microbiologie, de l’écologie, et de l’éco-toxicologie permettant de comprendre le rôle primordial des microorganismes dans le fonctionnement et la dynamique des écosystèmes naturels ou anthropisés, et de manière symétrique, d’évaluer les conséquences écologiques des perturbations du milieu sur les communautés microbiennes et sur les fonctions dont elles sont responsables.
Le rôle de la diversité taxinomique et fonctionnelle des communautés microbiennes dans le fonctionnement des écosystèmes naturels sera illustré au travers d’exemples dans des écosystèmes terrestres et aquatiques par rôle des microorganismes dans les transformations de différents composés et les conséquences sur les écosystèmes.
Les conséquences écologiques de perturbations d’origine anthropique (pollution, changement globaux) à l’échelle des communautés microbiennes seront présentés, ainsi que l’utilisation des microorganismes et de leurs capacités de dégradation, notamment des xénobiotiques, dans la protection des écosystèmes et la réhabilitation de sites contaminés par les activités industrielles. Les activités microbiennes impliquées dans ces activités de dépollution seront détaillées.
Les travaux pratiques permettront de construire et mettre en œuvre une démarche scientifique en réponse à une problématique, d’illustrer les concepts de résistance et de résilience, d’analyser la biodiversité et les conséquences de perturbation à l’échelle des communautés microbiennes.
Responsable : Agnès Richaume-Jolion
Pour plus d’informations, rendez-vous ici.
Responsable : Bernard Kaufman
Master 2 - Semestre 3
Approfondissement des connaissances en Génomique en Ecologie et Evolution.
Analyse bioinformatique des données de séquencage obtenues au S2 et communication des résultats dans l’UE Ateliers d’analyses en génomique environnementale.
Ouverture scientifique sur des grandes questions en écologie – séminaires de recherche, en anglais, par des spécialistes français et étrangers.
Présentation de la métagénomique / métatranscriptomique comme discipline émergente: portée, limites et challenges; et analyse de données.
Choix d’une UE parmi 2 options : Phylogénomique (reconstruction d’arbres, écologie évolutive) ou Biodiversité et Fonctionnement des Écosystèmes (écologie trophique et fonctionnelle).

Journée d’intégration en M1 GE: échantillonnage d’ADNe à Vallorcine en Haute-Savoie. Les ADNe prélevés dans le ruisseau du Loriaz permettent-ils de décrire la diversité de ce valon alpin ?
1/ Évolution moléculaire
– Introduction : l’évolution moléculaire avant et après la révolution génomique
– Avancées actuelles en évolution moléculaire : quelques exemples approfondis
– Évolution de la taille des génomes et écologie des organismes
– Les éléments transposables et leur impact sur le génome
– Origine et évolution des chromosomes sexuels des animaux et des plantes
– Variation de la composition en base génomes
2/ Phylogénie
– Alignement : revue des différentes méthodes d’alignement de séquences
– Distance : Méthodes d’estimation des distances évolutives (modèle markovien, modèle spécifique)
– Phylogénie : Méthodes de reconstruction phylogénétique en insistant sur le maximum de vraisemblance,
les méthodes bayésiennes et l’évaluation de la fiabilité des arbres
– Discussion des biais dans les reconstructions en fonction des modèles évolutifs utilisés
3/ Mini-projets par groupes d’étudiants
Responsable : Céline Brochier-Armanet
L’écologie cherche à comprendre les causes et les conséquences de la dynamique de la biodiversité. Cette discipline s’intéresse à la fois aux mécanismes de l’évolution des êtres vivants en réponse à leur environnement (et à ses modifications) mais également au rôle qu’ont ces organismes dans le fonctionnement des écosystèmes. L’objectif de cette UE est de couvrir les points chauds, les concepts novateurs et les approches originales des recherches actuelles en écologie. Cet enseignement reposera sur des interventions de spécialistes français et étrangers de réputation internationale dans le domaine.
Nous aborderons cette discipline aux différents niveaux d’organisation biologique. Nous nous attacherons plus particulièrement à décrire les mécanismes clés qui modèlent et structurent la biosphère (e.g. relations entre être vivants, dynamique des écosystèmes, relations diversité/écosystème) au niveau génomique, populationnel et écosystémique.
Enfin, nous replacerons l’écologie dans un point du vue sociétal, économique et politique en abordant les thématiques liées à l’ « ecosystem assessment » et en prenant en compte les enjeux géo-politiques liées aux ressources naturelles.
Responsable : Christophe Douady et Nicolas Goudemand
Cette UE a pour objectif de donner aux étudiants les outils pour comprendre ce que sont les approches de métagénomique et métatranscriptomique (approches non ciblées appliquées à l’ADN et ARN environnemental, respectivement) et quelles sont les méthodologies mises en oeuvre pour traiter ces gros jeux de données issues de séquençage haut-débit. Cette UE leur permettra d’appréhender la diversité des trois domaines du vivant, Archaea, Bactéries et Eucaryotes mais également celle du compartiment Viral. Les différentes étapes de traitement permettront aux étudiants d’aboutir à une analyse biologique et une interprétation tant en terme taxonomique (par l’étude de la fraction ribosomique) que fonctionnelle (par l’étude de la fraction ARN messagers).
Une partie du cours illustrera la puissance de ces approches méta- et la plus-value de l’étude des ADN et ARN environnementaux dont les avantages sont multiples, incluant une application à différents champs d’étude (agriculture, environnement, agroalimentaire, santé), une compréhension intégrée du fonctionnement des écosystèmes ou encore des reconstructions de l’histoire passée de certains écosystèmes. Nous aborderons également la puissance des approches de métagénomique pour la reconstruction de génomes de (micro)organismes dont nous ne disposons pas à l’heure actuelle de représentants cultivés, approche particulièrement pertinente dans des écosystèmes de type milieux extrêmes où la richesse est faible. Un bref rappel sera consacré à la comparaison avec les approches de métabarcoding dispensées en M1.
Les limites de ces techniques méta-omiques seront abordées, au travers notamment de discussions autour de la puissance de séquençage permettant ou non de décrire l’ensemble de la diversité génétique et fonctionnelle et le transcriptome d’une communauté en mettant l’accent sur de nouvelles technologies de type Illumina ou Nanopore permettant de pallier ces soucis. De la même façon, les difficultés liées à l’identification des (micro)organismes portant différentes fonctions d’intérêt seront abordées et seront mises en relief de la nécessité de mise en œuvre d’approches complémentaires.Seront abordés les enjeux liés au traitement bioinformatique tels que les notions de contamination des jeux de données, d’assemblage, d’identification de gènes, d’affiliation ou encore d’estimation d’abondance. En lien avec ce qu’ils auront acquis au S2 dans l’UE MADGT, les étudiants apprendront à analyser ces données méta-omiques en TP et TD afin d’estimer des taux d’expression ou encore de conduire des analyses d’expression différentielle dans des designs à plusieurs conditions.
Responsable : Mylène Hugoni
L’UE « Biodiversité et fonctionnement des écosystèmes » a pour objectif d’apporter des connaissances essentielles pour comprendre les mécanismes impliqués dans la dynamique de la biodiversité aux différentes échelles spatiales, ainsi que le rôle de la biodiversité dans le fonctionnement des écosystèmes. Le contenu de cette UE abordera de manière approfondie les points suivants :
Dynamique de la biodiversité
-
Mesure et analyse de la biodiversité aux différents niveaux hiérarchiques (du gène à la communauté),
-
Les différentes échelles spatiales de la biodiversité : gradients latitudinaux et altitudinaux, taille de niche
-
Liens entre biodiversité, perturbations et dynamique des écosystèmes
-
Impact des changements climatiques et conséquences des invasions biologiques
Rôle de la biodiversité sur le fonctionnement des écosystèmes
-
Liens biodiversité-fonction : cadre théorique
-
Rôle de la biodiversité dans les cycles des éléments et les flux de matière : organismes ingénieurs, construction de niche
-
Groupes fonctionnels
-
Interactions trophiques : cascades trophiques, régulation des ressources par les consommateurs (« top-down »), rétroactions des ressources sur les consommateurs (« bottom-up »)
-
Traits fonctionnels dans les communautés
Génomique comparative, évolution des séquences codantes et non codantes
Relation Genotype/Phénotype/Environnement
Epigénomique populationnelle et hérédité non génétique, étude de cas avec des données ChiPseq, epiRIL
Evolution des systèmes de reproduction
Etude des pangénomes, core genomes
Responsable : Cristina Heddi
Le projet tuteuré prépare à l’insertion en milieu professionnel (ex. recherche académique/finalisée, entreprises). Ce projet correspond au sujet de stage (qui peut être proposé par l’équipe pédagogique, par un organisme d’accueil ou par l’étudiant après concertation avec un organisme d’accueil). Le projet comprend une initiation à la recherche bibliographique et l’appropriation des concepts et outils nécessaires à la réalisation du stage de recherche. Le travail fait l’objet d’un compte rendu écrit (poster) et donne lieu à une présentation orale.
-
Gestion de projet et recherche bibliographique,
-
Encadrement tutoré.
L’objectif est la compréhension des mécanismes impliqués dans la dynamique de la biodiversité aux différentes échelles spatiales, ainsi que le rôle de la biodiversité dans le fonctionnement des écosystèmes. Les 2 points suivants seront abordés (CM 18h) :
Biodiversité
Mesure et analyse de la biodiversité aux différents niveaux hiérarchiques (du gène à la communauté),
Différentes échelles spatiales de la biodiversité : gradients latitudinaux et altitudinaux, taille de niche,
Liens entre biodiversité, perturbations et dynamique des écosystèmes,
Impact des changements climatiques et conséquences des invasions biologiques.
Fonctionnement des écosystèmes
Rôle de la biodiversité dans les cycles des éléments et les flux de matière : organismes ingénieurs, construction de niche,
Groupes fonctionnels,
Interactions trophiques : cascades trophiques, régulation des ressources par les consommateurs (« top-down »), rétroactions des ressources sur les consommateurs (« bottom-up »),
Traits fonctionnels dans les communautés.
Pour plus d’informations, rendez-vous ici.
Responsable : Jean-Paul Léna
Cette UE organisera l’analyse et la diffusion des données obtenues dans l’UE “Biologie moléculaire pour la génomique environnementale”. L’UE est constituée d’ateliers dont le contenu dépendra des jeux de données produits aux semestres précédents. Les ateliers auront pour objectifs d’organiser l’analyse des jeux de données et seront ponctués d’interventions sur des points méthodologiques (bioinformatiques, statistiques) mais également sur des éléments de communications (archivage, documentation, rapport, présentation orale). Les objectifs attendus de cette UE sont la prise d’autonomie, la mise en place de bonne pratique en bioinformatique, le retour vers les questions écologiques et la communication de ces résultats.
Responsable : Tristan Lefebure
Master 2 - Semestre 4
Le S4 sera consacré entièrement au stage de longue durée (5 mois), effectué en laboratoire de recherche ou en entreprise.
Stage de recherche
Le stage de recherche
Le stage en entreprise d’une durée de 4 à 6 mois permet une immersion dans le monde du travail dans le domaine de la gestion environnementale des espaces naturels et urbains. Au cours du premier semestre, chaque étudiant doit mener sa propre recherche de stage sur la base d’un cahier des charges établi avec les responsables du M2. Pour chaque étudiant, le sujet du stage et le choix de la structure d’accueil (un bureau d’études, une collectivité, un syndicat mixte, un conservatoire, un laboratoire, etc) devront être validés par le Comité de direction.
Débouchés
Débouchés et insertion professionnelle
Le parcours GE a pour objectif de former des chercheurs, des chercheuses et des expert.es capables de construire et de mener à bien un projet de génomique environnementale dans son entièreté : la formulation d’une problématique d’écologie, la mise au point d’un protocole d’échantillonnage, le choix des outils moléculaires, statistiques, et bioinformatiques et l’analyse, l’interprétation et la communication des résultats au regard de la problématique. Les possibilités d’emplois en sortie de master sont diversifiées car ces compétences intéressent différentes structures :
Doctorat en écologie, évolution, génomique…(p.ex. au sein de l’école doctorale E2M2)
Ingénieur.e sur des projets de recherche
Ingénieur.e sur des plateformes de génomique environnementale (publique ou privée)
Bureaux d’études en génomique environnementale ou environnement
Entreprise du biomédicale…
L’atout majeur du parcours Génomique Environnementale (GE) consiste en une approche globale, avec l’apprentissage des compétences permettant une approche par projet de résolution de questions d’écologie intégrant des outils de génomique.
Les compétences bio-informatiques acquises au cours de ce parcours seront fortement valorisables à l’issue du master, quel que soit le débouché choisi.
Inscription
Modalités d'inscription et d'admission
L’admission en M1 se fait via la plateforme Monmaster.
L’admission en M2 est prononcée par une commission d’admission propre au parcours et composée des membres de l’équipe pédagogique. L’admission est conditionnée à l’obtention d’un diplôme (M1, école d’ingénieur,..) dans le domaine de l’écologie ouvrant droit à une inscription en M2 et au dépôt d’un dossier complet.
La capacité d’accueil du M2 étant limitée (capacité d’accueil de l’équipe pédagogique, bassin d’emploi), l’admission sera liée à la qualité du dossier ainsi qu’au projet professionnel défendu par le candidat.
Constitution du dossier :
En cas de difficultés, contacter la scolarité l’UFR Biosciences :
scolarité.biosciences@univ-lyon1.fr
L’ensemble des documents à fournir pour l’inscription vous sera indiqué dans la procédure d’inscription. L’obtention du stage n’est pas obligatoire pour l’inscription, mais vivement conseillée. Les étudiants auront jusqu’en octobre pour trouver un laboratoire d’accueil pour ce stage. Nous attirons votre attention sur le fait qu’une lettre d’acceptation d’une équipe de recherche s’engageant à vous prendre en stage de recherche sera demandée.
Tout dossier incomplet (sauf pour les notes du semestre 2 de M1) sera refusé. Les notes du semestre 2 seront à faire parvenir au plus vite à l’UFR Biosciences.
N’hésitez pas à contacter les responsables du parcours pour vous aider dans votre recherche de stage.