
EEG@Lyon
Parcours Écologie, Évolution, Génomique
Le Master BEE parcours EEG «Écologie, Évolution, Génomique» accueillera sa 9ème promotion d’étudiants à la rentrée 2024. Vous trouverez toutes les informations nécessaires pour découvrir l’offre de formation, son fonctionnement, les modalités d’inscription et les débouchés.
Bonne lecture !
Responsable : Laurence Mouton
Laboratoire de Biométrie et Biologie Évolutive (LBBE)
UMR CNRS 5558
Bâtiment Mendel
43 boulevard du 11 Novembre 1918
69622 VILLEURBANNE cedex
contact: laurence.mouton@univ-lyon1.fr
Formation
Objectifs de la formation
Les mécanismes qui sous-tendent la diversité biologique et son évolution nécessitent d’appréhender la donnée biologique à différentes échelles de temps (échelle brève telle que celle de l’impact de l’homme sur les populations, à très étendue comme les temps géologiques), d’espace et de niveaux hiérarchisés d’organisation (séquences, gènes, génomes, organismes, populations, communautés, écosystèmes). Cette complexité requiert des approches expérimentales complémentaires et multidisciplinaires (ou interdisciplinaires) et génère de nouveaux concepts et données nécessitant une grille de lecture et des méthodes appropriées.
L’objectif du parcours « EEG » du Master BEE est de former à et par la recherche des spécialistes de la biologie évolutive qui possèdent une maîtrise conceptuelle et expérimentale des mécanismes et processus de l’écologie et de l’évolution.
Compétences et savoir-faire visés à l’issue de la formation :
-
Capacité de synthèse,
-
Démarche intégrative,
-
Restitution de résultats scientifiques (orale et écrite),
-
Conduite d’un projet scientifique,
-
Travail en équipe
Originalités et forces de la formation EEG :
-
Accent mis sur l’interdisciplinarité : une nécessité pour aborder les questions complexes à différents niveaux d’organisation.
-
Formation adossée à des structures de recherche reconnues nationalement et internationalement. Ces laboratoires constituent un large vivier d’équipes d’accueil pour le stage du M2.
-
Diversité des interlocuteurs locaux allant des méthodologistes (mathématiciens, informaticiens, algorithmiciens, statisticiens, modélisateurs…) aux expérimentateurs à différentes échelles et travaillant sur une grande diversité de modèles biologiques.
Master 1 - Semestre 1
Pour plus d’informations sur le premier semestre du M1, rendez-vous ici.
Master 1 - Semestre 2
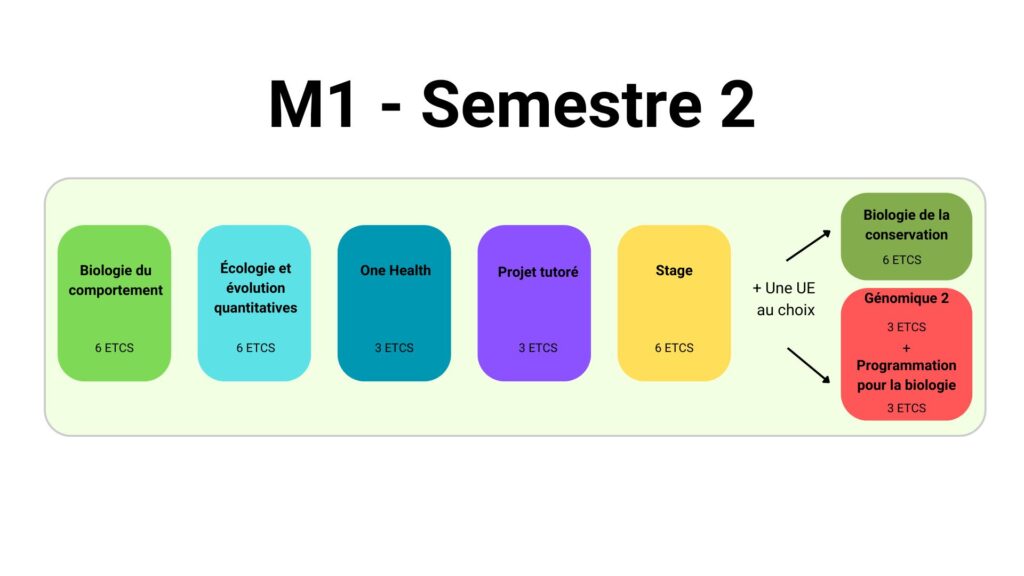
Le semestre 2 (faisant suite au semestre 1) se compose de 6 UE dont une pour laquelle deux choix sont possibles (« Biologie de la conservation » ou « Génomique 2 et Programmation pour la biologie »). Ce semestre se termine par un stage de deux mois dans un laboratoire de recherche en France ou à l’étranger. Ce stage peut être effectué au sein d’un organisme public ou privé. Le sujet de stage doit être en lien avec le projet professionnel de l’étudiant.e et doit lui permettre de mobiliser les compétences acquises au cours de sa formation.
Cette UE a pour objectif de présenter au travers des questions de Biologie Evolutive, différents modèles statistiques et modèles dynamiques permettant d’analyser des données issues de la littérature scientifique. Cette UE sera l’occasion pour les étudiants de répondre de manière concrète à des problématiques actuelles dans les domaines de l’écologie, de la génétique et de la phylogénie.
Elle se décompose en trois blocs :
-
Le premier bloc traite des questions liées aux processus évolutifs, notamment de l’évolution des génomes (modélisation par des chaînes de Markov en temps discret et continu). La prise en compte l’inertie phylogénétique dans les processus évolutifs au niveau interspécifique sera décrite via l’analyse comparative.
-
Le deuxième bloc se focalisera sur les données issues de suivis temporels en écologie (e.g. suivi des comportements individuels, analyse des changements de profils chimiques, …) et sur les données d’écologie spatiale (e.g. distribution spatiale des individus et de leurs ressources…). Pour aborder ces questions, les notions d’autocorrélation spatiale, dans sa forme exploratoire (une variable ou un ensemble de variables et une carte) et d’autocorrélation temporelle (modèle linéaire mixte, introduction au modèle linéaire généralisé) seront abordées.
-
Prendre en compte les aspects écologiques et évolutifs qui sous-tendent la dynamique des populations nécessitent de maîtriser des outils de modélisation (e.g., modèles épidémiologiques, modèles d’interaction de type prédateur/proie, modèles de Leslie). Ce bloc sera aussi l’occasion d’initier les étudiants aux modèles de survie.
L’UE « One Health » est un programme innovant et transdisciplinaire qui vous permettra de travailler dans plusieurs disciplines afin de relever des défis complexes en matière de santé unique, c’est-à-dire à l’interface entre santé humaine, animale et des écosystèmes. En effet, la population humaine devrait passer de 7,5 à 9,7 milliards d’individus d’ici à 2050. Toutes les sociétés sont donc actuellement confrontées au grand défi de maintenir les populations humaines croissantes bien nourries et en bonne santé (notamment dans les zones urbaines où plus de 5 milliards de personnes vivront d’ici 2030) tout en ne mettant pas en péril la biodiversité et les ressources naturelles de la planète. La question de la santé ne peut plus être réduite aujourd’hui à l’humain. Elle doit être abordée dans une perspective beaucoup plus large face à l’augmentation rapide de l’empreinte humaine dans pratiquement tous les écosystèmes de la planète : influences anthropiques (par exemple, urbanisation, agriculture, pollution des sols, de l’eau et de l’air), réchauffement de la planète (par exemple, augmentation de la fréquence des phénomènes climatiques extrêmes) ou perte de biodiversité (par exemple, dégradation des habitats, propagation des espèces envahissantes et des espèces porteuses d’agents pathogènes).
L’enseignement commencera par un cours magistral de 3h exposant le concept de One Health, son histoire, son importance pour la santé humaine et les grands exemples qui illustrent son intérêt. Il sera suivi de deux grandes parties illustrant par l’exemple la démarche présentée :
– la résistance aux antibiotiques – les invasions biologiques
Responsable : David FouchetCette UE a pour vocation de faire travailler les étudiants en petits groupes avec une approche professionnelle sur des projets en lien direct avec leur formation.
Les projets sont structurés autour du triptyque : élaboration d’un cahier des charges, développement et rendu des livrables. Ils permettent aux étudiants de développer les compétences conceptuelles, méthodologiques et pratiques mises en œuvre au cours de leur formation, ainsi que la maîtrise des outils et des approches requis pour le travail en équipe / collaboratif (e.g. cahier de laboratoire, partage de documentation).
Pour plus d’informations, rendez-vous ici.
Responsable : Annabelle Haudry
Cette UE traite du versant biologique de la conservation de la biodiversité, tant au niveau de la viabilité des populations que de la diversité des stratégies biologiques. L’approche de cette UE est donc plutôt espèce centrée, les approches de conservation et gestion des milieux étant plutôt abordées dans les trois autres UE de semestre 2. Dans le cadre de cette UE, il s’agira donc d’intégrer les acquis de différentes disciplines (démographie, génétique de populations, écologie comportementale, biologie évolutive) pour documenter le statut de conservation des populations et prédire leur altération consécutive de la fragmentation des habitats, de l’introduction d’espèces allochtones, ou de prélèvements abusifs. Les conditions de restauration d’une population sont analysées en particulier à la lumière de l’analyse des structures spatiales des paysages (notion de connectivité).
Les travaux dirigés et les travaux pratiques visent à donner aux étudiants une compétence opérationnelle quant aux outils utilisés en biologie de la conservation : approche intégrative en gestion des milieux et des écosystèmes (indicateurs de changements écologiques, indices de fonctionnement), approche démographique pour l’analyse de viabilité des populations (estimation des paramètres démographiques et modèles de projection) et approche génétique en conservation (hybridation, structuration et diversité génétique des populations, génétique du paysage).
Responsable : Jean-Paul LenaDynamique des génomes. Taille des génomes, éléments transposables, duplications.
Contribution des transfert horizontaux à l’évolution des génomes eucaryotes
Recombinaison, sélection et évolution du génome : conséquences de la recombinaison et de l’absence de recombinaison dans l’évolution du génome. Interaction entre recombinaison et sélection.
Reconstruction de l’histoire évolutive des populations, introduction au coalescent, impact de la sélection, tests de détection de sélection.
Responsable : Annabelle HaudryL’objectif de cette unité d’enseignement est d’assurer aux biologistes une certaine autonomie en bioinformatique pour le développement d’outils spécifiques à leur problématique biologique et au traitement bioinformatique de leurs données.
L’UE est uniquement sous la forme d’un enseignement pratique devant ordinateur. Aux aspects pratiques de la programmation pour la manipulation de données, un objectif est d’illustrer l’intérêt interdisciplinaire de l’informatique pour traiter des problématiques biologiques complexes.
La problématique biologique traitée sera la génétique des populations, par la conception et la simulation de dynamiques évolutives mettant en jeu des mécanismes aussi réalistes que possible, à confronter avec statistiques connues.
Responsable : Laurent GueguenPour plus d’informations, rendez-vous ici.
Responsable : Bernard Kaufmann
Master 2 - Semestre 3
La formation dispensée fera appel à des experts scientifiques nationaux et internationaux. Des cycles de conférences interdisciplinaires sur des thématiques au front de la recherche scientifique permettront aux étudiants de combiner leurs compétences en écologie et génomique évolutives, et méthodologie (UE AIEE Concepts) et de juger les apports de l’interdisciplinarité.
2 UEs dédiées à l’insertion professionnelle sont proposées (Anglais et Projet tutoré). Cette dernière UE vise à aider les étudiants à s’approprier leur problématique de recherche, la bibliographie propre au sujet et à prendre en main des méthodes (e. g. statistiques) à mettre en œuvre lors du stage du semestre 2.
3 UEs optionnelles complètent la formation académique et permettent à chacun d’individualiser son parcours de formation. Par exemple, si la formation souhaitée se veut plus proche de l’écologie, l’étudiant pourra choisir Biodiversité et Fonctionnement des Écosystèmes et Statistiques Bayésiennes. Si une formation plus transversale est recherchée, l’étudiant peut opter par les UEs Statistiques Bayésiennes et Phylogénomique et Evolution Moléculaire.
Pour le choix des options, il est conseillé de discuter avec le responsable de la formation et votre encadrant de master afin de choisir celles qui correspondent le mieux à vos besoins futurs et votre projet professionnel.

Préambule : Cette UE fait appel à des notions et concepts complexes qui nécessitent un investissement des étudiant.e.s dans la lecture d’articles scientifiques pour poursuivre la réflexion initiée par les intervenant.e.s. Il est conseillé aux étudiant.e.s de demander aux responsables des thèmes les lectures les plus appropriées pour chaque thème.
Objectifs généraux :
Les étudiant.e.s seront confronté.e.s, au cours de 4 cycles de conférences à des approches trans- et inter-disciplinaires sur des questions majeures et actuelles en écologie, évolution et génomique. Lors des conférences seront présentées comment, dans un même cadre conceptuel, la prise en compte de différents niveaux d’organisations, de diverses approches (disciplinaires et méthodologiques) augmente notre compréhension du vivant et font émerger des axes de recherche pour le futur.
Organisation de l’UE :
Thème 1 : Epistémologie dans les sciences de l’évolution (8h)
Ce thème vise à développer une culture générale et une vision critique sur les pratiques scientifiques, notamment dans le domaine des sciences de l’évolution. Les enseignements seront assurés par Fabienne Paulin (didacticienne, S2HEP, Lyon1) et Sylvain Charlat (biologiste de l’évolution, LBBE, Lyon1). Les interventions porteront à la fois sur une synthèse des théories et concepts majeurs de l’épistémologie, et sur leurs articulations spécifiques avec la théorie de l’évolution. Trois livres, entre autres, peuvent permettre d’approfondir ce cours : « What is this thing called science? » de Chalmers (également disponible en français), « De la vérité dans les sciences » de Barrau et « Epistémologie de la biologie » de Dupouey.
Thème 2 : Approche intégrative du processus de sénescence
Les probabilités de survivre et de se reproduire des individus déclinent au cours de la vie. Ce processus de sénescence, bien que quasi-universel dans le vivant, n’en demeure pas moins extrêmement variable, que ce soit entre les espèces, les populations ou les individus. L’étude de la sénescence nécessite aujourd’hui une approche intégrée ayant pour objectifs de quantifier cette variabilité à différentes échelles taxonomiques, de déterminer ses causes évolutives et ses conséquences (notamment en termes de santé publique et de démographie), et d’identifier les mécanismes physiologiques et génétiques qui régissent la diversité des trajectoires observées dans la nature. Quatre interventions de 2h chacune illustreront l’importance de l’interdisciplinarité dans l’étude de la sénescence :
– Théories évolutives de la sénescence (Jean-François Lemaitre, LBBE) – Modélisation de la sénescence (Jean-Michel Gaillard, LBBE) – Mécanismes génétiques et physiologiques de la sénescence en milieu naturel (Yann Voituron, LEHNA) – Approches évolutionnistes des maladies de la sénescence (Dominique Pontier, LBBE)
Thème 3 : Biodémographie évolutive
Comprendre comment les changements globaux influencent les populations sauvages nécessite une approche reliant dynamiques évolutives et dynamiques démographiques. Le volet « Biodémographie Evolutive » vise à introduire les concepts et les outils méthodologiques relatifs à l’étude de ces dynamiques éco-évolutives dans les populations naturelles. Ce volet se compose d’interventions (Jean-Michel Gaillard et Marlène Gamelon, CNRS, LBBE) et de présentations réalisées par les étudiant.e.s de quelques-uns des articles scientifiques fondateurs de cette discipline.
Thème 4 : Plasticité Phénotypique, Evolution et Épigénétique
La plasticité phénotypique est définie comme la capacité d’un génotype à produire différents phénotypes en fonction des conditions environnementales. Si l’importance de la plasticité phénotypique comme source de variabilité phénotypique est indéniable, des interrogations persistent sur l’importance de la plasticité phénotypique dans les processus évolutifs. L’objectif de cette UE sera donc de clarifier la notion de plasticité phénotypique, son évolution, son rôle évolutif ainsi que les mécanismes génétiques et épigénétiques sous-jacents.
Responsable : Emmanuel Desouhant & Laurence MoutonComprendre comment la sélection écologique génère des changements dans les traits phénotypiques est un objectif majeur de la biologie de l’évolution. Ces changements sont le résultat de facteurs externes (par exemple, des changements dans les conditions environnementales) et de facteurs internes (par exemple, les changements dans les corrélations génétiques ou l’architecture génétique sous-jacente aux traits phénotypiques).
Dans ce contexte, seront abordés dans cette UE les concepts qui sous-tendent les trajectoires évolutives à différents niveaux d’organisation du vivant ainsi que les méthodes permettant d’analyser et de prédire ces trajectoires :
-
Architecture génétique et génomique et trajectoire évolutive
-
Plasticité phénotypique transgénérationnelle
-
Early life conditions et leurs conséquences, personnalité animale
-
Adaptation locale sous différents régimes de sélection
-
Modélisation : dynamique adaptative
La biologie évolutive et l’écologie partagent les mêmes objectifs : décrire la variabilité observable dans les systèmes naturels et en comprendre les facteurs explicatifs. Dans ce contexte, tous acquièrent des données, expérimentales ou in situ.Partant de cas concrets et à l’aide du logiciel R, l’objectif de cet enseignement est de montrer comment la statistique s’implique dans la recherche d’une information structurant les données. Les thèmes abordés :
-
Modèles descriptifs permettant de croiser deux tableaux de données contenant des informations de nature différente (e.g., ACP inter et intra-classe, analyse canonique des redondances, analyse de coinertie, classification),
-
Utilisation de modèles probabilistes pour analyser des jeux de données à échelles de variation multiple (e.g., modèle linéaire mixte, modèle linéaire généralisé mixte),
-
Utilisation de critères d’information pour sélection un ensemble de covariables explicatives.
Génomique comparative, évolution des séquences codantes et non codantes Relation Genotype/Phénotype/Environnement Epigénomique populationnelle et hérédité non génétique, étude de cas avec des données ChiPseq, epiRIL Evolution des systèmes de reproduction Etude des pangénomes, core genome
Responsable : Cristina HeddiL’enseignement est dispensé par le Service Commun d’Enseignement des Langues de Lyon 1 en vue de l’obtention du niveau B1 (http://scel.univ-lyon1.fr). L’objectif est de maîtriser la réaction scientifique d’un résumé et d’un article scientifique, la construction d’un poster scientifique pour communiquer des résultats scientifiques.
Le parcours du master a été sélectionné pour que les étudiants accèdent à un niveau de certification plus élevé. Ce dispositif va se mettre en place dans les mois à venir.
Responsable : Nathalie DourlotLe projet tuteuré prépare à l’insertion en milieu professionnel (ex. recherche académique/finalisée, entreprises). Ce projet correspond au sujet de stage (qui peut être proposé par l’équipe pédagogique, par un organisme d’accueil ou par l’étudiant après concertation avec un organisme d’accueil). Le projet comprend une initiation à la recherche bibliographique et l’appropriation des concepts et outils nécessaires à la réalisation du stage de recherche. Le travail fait l’objet d’un compte rendu écrit (poster) et donne lieu à une présentation orale.
-
Gestion de projet et recherche bibliographique,
-
Encadrement tutoré.
Pour plus d’informations, rendez-vous ici.
Responsable : Laurence Mouton
L’objectif est la compréhension des mécanismes impliqués dans la dynamique de la biodiversité aux différentes échelles spatiales, ainsi que le rôle de la biodiversité dans le fonctionnement des écosystèmes. Les 2 points suivants seront abordés (CM 18h) :
-
Biodiversité
-
Mesure et analyse de la biodiversité aux différents niveaux hiérarchiques (du gène à la communauté),
-
Différentes échelles spatiales de la biodiversité : gradients latitudinaux et altitudinaux, taille de niche,
-
Liens entre biodiversité, perturbations et dynamique des écosystèmes,
-
Impact des changements climatiques et conséquences des invasions biologiques.
-
-
Fonctionnement des écosystèmes
-
Rôle de la biodiversité dans les cycles des éléments et les flux de matière : organismes ingénieurs, construction de niche,
-
Groupes fonctionnels,
-
Interactions trophiques : cascades trophiques, régulation des ressources par les consommateurs (« top-down »), rétroactions des ressources sur les consommateurs (« bottom-up »),
-
Traits fonctionnels dans les communautés.
-
Objectif :
Initier les étudiants à l’inférence bayésienne, ainsi qu’aux outils techniques nécessaires à son implémentation. A l’issue de ce module, l’étudiant sera capable de comprendre et d’expliciter les différences entre l’inférence bayésienne et l’inférence fréquentiste, ainsi que les forces et limites de chacune, d’interpréter l’estimation des paramètres d’un modèle dans un cadre bayésien et de valider ce modèle.
Programme :
Théorie de l’inférence bayésienne :
-
Principe de l’inférence bayésienne
-
Méthode de l’inférence bayésienne : solution explicite, algorithmes MCMC (Metropolis-Hastings et Gibbs sampling)
-
Choix des lois à priori
-
Estimation des paramètres d’un modèle
-
Validation et comparaison de modèles
Chacune des parties sera illustrée par des applications concrètes.
Mise en œuvre avec les outils de type « BUGS » :
-
Formalisation d’un modèle sous la forme d’un graphe acrylique dirigé
-
Introduction aux outils de type « BUGS »
-
Prise en main du logiciel JAGS à partir d’exemples simples
Ce module combine des cours théoriques, pendant lesquels seront présentés les concepts généraux de la phylogénie moléculaire (comment reconstruire les phylogénies; comment les utiliser pour comprendre les processus d’évolution des séquences génétiques; quelles utilisations en sont faites aujourd’hui en bioinformatique et plus généralement en biologie) et des travaux pratiques utilisant un logiciel spécialement dédié à la phylogénie moléculaire, RevBayes (revbayes.github.io), permettant de construire des modèles statistiques variés et de les appliquer à des données moléculaires réelles. À l’issue de ce module, l’étudiant.e aura acquis les connaissances et compétences de base pour reconstruire des phylogénies et comprendre le sens et les propriétés des méthodes statistiques qui sont employées dans ce domaine fondamental de la bioinformatique.
1/ Évolution moléculaire
– Introduction : l’évolution moléculaire avant et après la révolution génomique
– Avancées actuelles en évolution moléculaire : quelques exemples approfondis
– Évolution de la taille des génomes et écologie des organismes
– Les éléments transposables et leur impact sur le génome
– Origine et évolution des chromosomes sexuels des animaux et des plantes
– Variation de la composition en base génomes
2/ Phylogénie
– Alignement : revue des différentes méthodes d’alignement de séquences
– Distance : Méthodes d’estimation des distances évolutives (modèle markovien, modèle spécifique)
– Phylogénie : Méthodes de reconstruction phylogénétique en insistant sur le maximum de vraisemblance, les méthodes bayésiennes et l’évaluation de la fiabilité des arbres
– Discussion des biais dans les reconstructions en fonction des modèles évolutifs utilisés
3/ Travaux Pratiques
– simulation et inférence phylogénétique
– exploration de différents modèles d’évolution des séquences d’ADN
– modéliser les variations de vitesse d’évolution entre gènes, entre sites.
– les modèles phylogénétiques à codons
– datation moléculaire
4/ Mini-projets individuels: dans la continuité des travaux pratiques
Responsable : Céline Brochier-ArmanetMaster 2 - Semestre 4
Il est consacré à un stage obligatoire de longue durée (30 ECTS). Ce stage fait l’objet de la rédaction d’un rapport (voir organisation et évaluation) et d’une soutenance orale
Stage de recherche
Le stage de recherche et les équipes d'accueil
Le stage de recherche
Le stage permet une immersion dans le monde de la recherche. Pour chaque étudiant, le sujet du stage et le choix du laboratoire devront être validés par les responsables du M2.
Demandez des conseils à la responsable du M2, consultez les équipes au sein des structures d’accueil.
Voici quelques exemples d’offres de stages possibles :
Offres de stage
– Etude des voies de signalisation impliquées dans la régulation de la dynamique symbiotique chez un insecte ravageur des cultures
– Protection intégrée des céréales à paille. Elaboration de protocoles d’expérimentations en conditions contrôlées sur les insectes vecteurs de viroses
– Does metabolic scaling optimize fitness? A test based on Dynamic Energy Budget theory.
– Annotation et analyse évolutive des gènes responsables du parfum de la lavande laineuse
– Caractérisation de l’évolution du fardeau génétique chez des insectes envahissants, par des approches de génomique des populations
– Contrôle du ravageur invasif Drosophila suzukii par interférence reproductive interspécifique
– Effets de la diversité intra- et inter-spécifique et des phases du cycle de vie sur la dynamique du recrutement des communautés microbiennes chez Pisum spp.
– Genomic determinants of adaptation to host species in a population of the phytopathogenic fungus Zymoseptoria tritici
– Le silure aux abords des ouvrages de franchissement en grandes rivières : facteurs biotiques et abiotiques de présence et analyse comportementale
– Bases génétiques de l’adaptation locale et vulnérabilité génomique chez l’ancêtre sauvage du navet, Brassica rapa
– Recherche des déterminants génomiques de la diversité phénotypique chez les mammifères
– Does regeneration re-use developmental gene regulatory networks?
– Deciphering the origins of phototrophy in Pseudomonadota (Proteobacteria) using genomic data
– Apprentissage chez un insecte ravageur, Drosophila suzukii : de l’importance du contexte
– Contraintes du milieu physique sur la structure et la stabilité des modules trophiques aquatiques
– Méta-analyse du rôle de la composition des odeurs de fleurs dans les interactions plantes-pollinisateurs
– Phylogenetic inference in recombinant sequences using Hidden Markov Models. Application to the study of gene conversion dynamics in paramecium
– Analyse statistique du compromis entre croissance et reproduction chez les Chênes
– Influence des communautés fongiques sur l’absorption de métaux lourds par la plante : conséquences pour la mise en place des techniques alternatives
– Patterns of senescence in the Asian elephant (Elephas maximus)
– Characterization of transposable element expression patterns and their role in genomic stability during speciation in Ficedula flycatchers
– Effets génétiques indirects vs effets environnementaux chez une espèce de mammifère social : la marmotte Alpine
– Modeling the effects of plant-fire and plant-nutrient feedbacks on succession dynamics and ecosystem functioning
– Investigation of the role of splicing variants in the context of speciation
– Dynamique évolutive des structures génomiques en une et trois dimensions
– What are the most important factors shaping home range size in Felids globally ?
– The role of CpG islands in recombination rate regulation in a passerine bird
– Detection of balancing selection in Ficedula flycatchers
– Influence de la bactérie Wolbachia sur les limites thermiques du ravageur de cultures Drosophila suzukii
– Étude de l’héritabilité et de la sélection sur le comportement de dispersion dans une population naturelle de lézard vivipare
– Quantification du processus d’évolution régressive à l’échelle génomique lors de la colonisation du milieu souterrain
– Évaluation de la résistance du pois aux pucerons au champ et en conditions contrôlées
– Response of plant metabolome to rising temperatures in aquatic plants of the sub-Antarctic region
– Des souris et des hommes : Phéno/Génotypage nouvelle génération pour comprendre l’adaptation de la souris à l’anthropisation
– Paysages énergétiques et utilisation de l’habitat: le case du cerf en Corse
– The role of DNA methylation in the symbiosis between a cereal weevil and its endosymbiotic bacteria
– Deciphering the molecular dialogue between the mutualistic symbiosis between the cereal weevil Sitophilus oryzae and the endosymbiotic bacteria Sodalis pierantonius
– Dissecting the developmental origin and formation of the symbiont bearing-organ in the insect Sitophilus spp
– Fonction(s) des peptides antimicrobiens dans la symbiose mutualiste entre le charançon des céréales Sitophilus oryzae et la bactérie endosymbiotique Sodalis pierantonius
Les équipes d’accueil
Principales structures d’accueil rattachées au M2 EEG :
UMR CNRS 5558 Laboratoire de Biométrie et Biologie Evolutive, LBBE
(http://lbbe.univ-lyon1.fr/)

UMR CNRS 5023 Laboratoire d’Ecologie des Hydrosystèmes Naturels et Anthropisés, LEHNA
(http://umr5023.univ-lyon1.fr/)

UMR INRA 203 Laboratoire de Biologie Fonctionnelle Insectes et Interactions, BF2I
(http://bf2i.insa-lyon.fr)
Autres équipes françaises ou étrangères :
INRA Sophia Antipolis ; équipe BPI
Station d’Ecologie Expérimentale du CNRS à Moulis
UMR 7261 CNRS, Institut de Recherche sur la Biologie de l’Insecte
UMR 406 INRA-UAPV Abeilles et Environnement
…

Débouchés
Débouchés et insertion professionnelle
L’insertion professionnelle est au cœur de la formation. Elle est assurée via une formation approfondie à la communication scientifique, à la gestion de projet et à la réalisation d’un stage longue durée.
Notre formation prépare :
à l’entrée dans les écoles doctorales (ED) dont le champ scientifique couvre l’écologie, la biologie de l’évolution et la biodiversité (par exemple l’ED 341 E2M2 )
aux métiers de la recherche dans les secteurs publics (Universités, Muséum, CNRS, INRA, IRD, IRSTEA, CIRAD, Grandes Ecoles) et privé (par exemple, protection des plantes et environnement, collection et gestion des ressources biologiques, biovigilance, santé humaine et animale),
aux métiers faisant appel à une expertise scientifique en écologie et en évolution (PME-PMI, collectivités territoriales, Etat, …).
Diverses offres de thèses et d’emploi se trouvent sur les sites suivants :
· Société Française d’Ecologie (SFE)
· Fondation pour la recherche sur la Biodiversité
· Sites de diffusion et associations :
Inscription
Modalités d'inscription et d'admission
L’admission en M1 se fait via la plateforme Monmaster.
L’admission en M2 est prononcée par une commission d’admission propre au parcours et composée des membres de l’équipe pédagogique. L’admission est conditionnée à l’obtention d’un diplôme (M1, école d’ingénieur,..) dans le domaine de l’écologie ouvrant droit à une inscription en M2 et au dépôt d’un dossier complet.
La capacité d’accueil du M2 étant limitée (capacité d’accueil de l’équipe pédagogique, bassin d’emploi), l’admission sera liée à la qualité du dossier ainsi qu’au projet professionnel défendu par le candidat.
L’étudiant devra obligatoirement solliciter un entretien avec le responsable du parcours avant l’inscription (ces entretiens peuvent se réaliser par téléphone ou via une visioconférence (telle que Webex)). En l’absence de prise de contact au moment de la commission d’admission, le dossier sera refusé ou au mieux placé en liste d’attente.
Constitution du dossier :
En cas de difficultés, contacter la scolarité l’UFR Biosciences :
scolarité.biosciences@univ-lyon1.fr
L’ensemble des documents à fournir pour l’inscription vous sera indiqué dans la procédure d’inscription. L’obtention du stage n’est pas obligatoire pour l’inscription, mais vivement conseillée. Les étudiants auront jusqu’en octobre pour trouver un laboratoire d’accueil pour ce stage. Nous attirons votre attention sur le fait qu’une lettre d’acceptation d’une équipe de recherche s’engageant à vous prendre en stage de recherche sera demandée.
Tout dossier incomplet (sauf pour les notes du semestre 2 de M1) sera refusé. Les notes du semestre 2 seront à faire parvenir au plus vite à l’UFR Biosciences.
N’hésitez pas à contacter le responsable du parcours pour vous aider dans votre recherche de stage.